Apilactobacillus kunkeei とその近縁株の分類に関する新たな発見
山口大学 大学研究推進機構の前野 慎太朗 助教と東京農業大学 応用生物科学部 食品安全健康学科の遠藤 明仁 教授の共同研究により、ミツバチの消化管内で主要な菌のひとつとされるフルクトフィリック乳酸菌
Apilactobacillus kunkeei(
A. kunkeei)およびその近縁株の分類における課題が明らかになりました。近年主流の分類手法である平均ヌクレオチド類似度(ANI)やデジタルDNA-DNAハイブリダイゼーション(dDDH)を用いた分類方法が、
A. kunkeeiとその近縁株において一貫した結果を示さず、正確な分類及び同定が極めて困難であることが明らかにされました。
従来、細菌分類においてこのような問題は報告されていませんでしたが、複数の分類手法が普及している現在、他の細菌種でも同様の問題が将来的に発生する可能性があることが示唆されます。近年、ゲノム解析を用いた菌種同定法が広く利用されていますが、今回の発見は従来の菌種分類に限界があることを示唆しており、分類基準に関する議論が求められます。また今回の研究成果は、生物の「種」の概念を考えるうえで基礎となる重要な結果を示しています。本研究の成果は、2024年11月4日(英国時間)にELSEVIER「
Systematic and Applied Microbiology」誌で公開されました。
研究成果のポイント
| ● A. kunkeei関連株の分類において、既存のゲノム配列を用いた複数の菌種同定ツール(ANI、dDDH、GTDB-Tk)の間で矛盾した分類結果を示しました。このことは細菌分類の正確性や妥当性に影響を及ぼす可能性があります。 |
| ● GTDB-Tkが採用する「複数系統群の基準株と比較し、最も高い相同性を示す菌種に分類する」というルールを他のツールにも適用することで、解析の結果、複数の分類群に割り当てられた場合でも混乱が少なく分類できる可能性があることを示唆しました。 |
| ● 乳酸菌の正確な同定は、乳酸菌の食品利用における安全性の評価などの面において非常に重要であり、本研究は、ゲノム解析に基づく細菌分類のルールの改善に向けた非常に重要な知見を提供しています。 |
研究の背景
細菌の分類において、従来はDDH法が使用されてきましたが、実験の難易度やコストの高さから、近年では全ゲノム解析に基づく菌種分類が主流です。その中でも、ANIおよびdDDHは細菌の種レベルでの識別における基準として一般的に使用されており、通常はANIの95%、dDDHの70%という閾値が識別の基準とされています。
一方、
A. kunkeeiはミツバチの消化管内細菌として発見されたフルクトフリック乳酸菌で、この菌種の分類には、これまでにANIによる分類が困難である可能性が示唆されていました。本研究は、
A. kunkeeiおよびその近縁株における主流な解析ツールを用いて、それらの結果を比較し、検証することを目的としています。
研究の概要
本研究では 71株の
Apilactobacillus属株を対象に、3つの主要なゲノム解析ツールであるANI、dDDH、GTDB-Tkを用いて各菌株の分類を試み、その結果を比較しました。ANIおよびdDDHはBLASTを用いた相同性や、類似した遺伝領域の計算を通じて分類を行いますが、
A. kunkeeiおよびその近縁株においては、これらのツールの結果が一致しない場合がありました。また、GTDB-Tkは、試験菌株と複数の分類群の基準株との間で最も相同性が大きい分類群に分類する行う手法であり、ANIやdDDHにより閾値付近の値を取る
A. kunkeeiとその近縁株においては有用な解釈方法である可能性を見出しました。本研究では、複数の分類手法を比較し、その矛盾点を明確にしたことで、分類手法の課題を明らかにしました。
研究の成果
本研究の成果は、菌種分類の課題を明らかにしており、今後の菌種分類のルールを検討する際の重要な知見となる可能性を示しています。本研究では
A. kunkeeiとその近縁株を例に、ゲノム解析を行った結果、次の知見を得ました。
・複数の分類方法が使用されることによる問題
ANIやdDDHは、それぞれ異なるアルゴリズムと閾値を用いており、
A. kunkeeiおよびその近縁株においては分類結果が異なる菌株が存在することが明らかとなりました。複数の分類群にまたがる場合には、ANI、dDDHのいずれを使用するにしてもGTDB-Tkのように基準株に対して最も値が大きい分類群に分類するというルールを適用することで、異なる種にまたがる分類の曖昧さを解消する可能性が期待できます。
・新たな細菌分類の基準確立に向けた提案
現在のところ複数の分類群にまたがる場合どのように分類するべきかという決定事項はなく、議論も十分にされてきませんでした。本研究でのGTDB-Tkで採用されている、より近い分類群に分類するというルール(図1)を、他のツールにも導入することを提案しました。また、複数の分類ツール間での結果の不一致は、そもそも複数のツールが存在していることが原因と考えており、より正確で、多くの研究者の理解が得られる分類方法の確立に向けて今後の議論を進めるための重要な知見を提供しました。
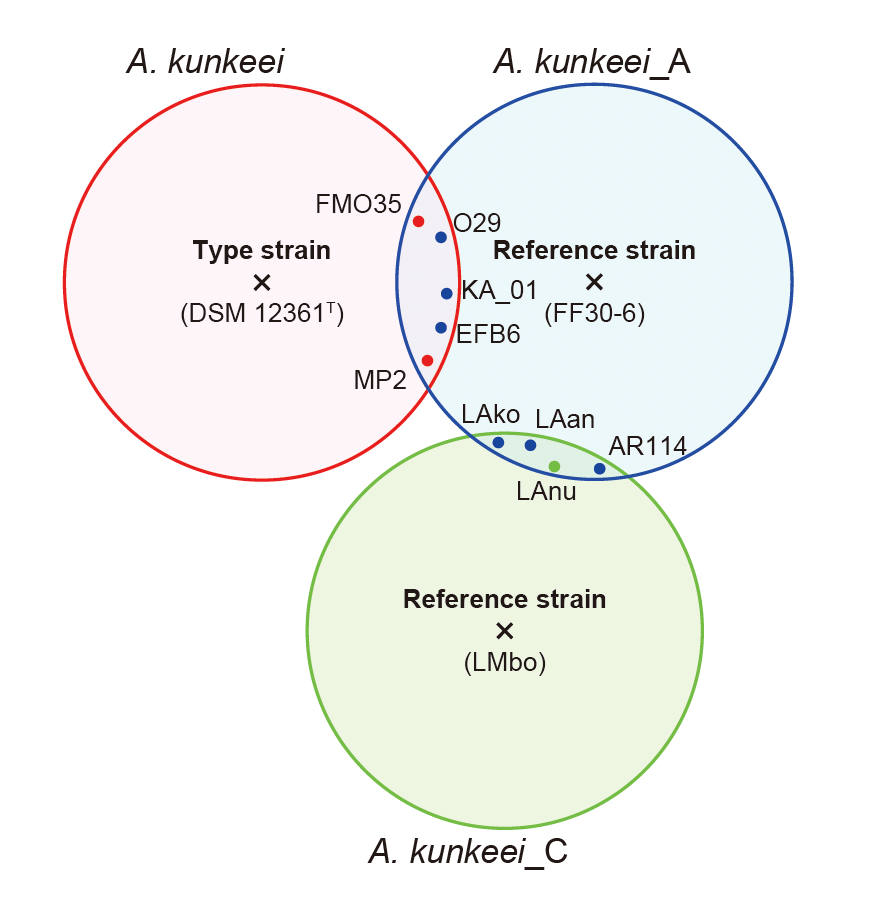
図1 複数の分類群にまたがる場合に最も近い分類群に分類するGTDB-Tk で用いられるルール
研究の展開
今回の提案を踏まえ、ANIやdDDHにもGTDB-Tkのアプローチが導入されることで、分類結果の一部の矛盾が解消される可能性が示唆されました。これにより、微生物学分野における分類基準の標準化が促進されることが期待されます。また、
A. kunkeeiおよびその近縁株以外の、分類が困難とされる他の細菌群にも、本研究の結果から分類が見直される潮流ができることが予想されます。
さらに、乳酸菌の分類及び同定は、乳酸菌の食品利用の際の安全性評価において非常に重要な指標となります。そのため、その分類及び同定を正確に行うことは乳酸菌の産業利用のために重要です。今後は本研究の成果に基づき、より信頼性が高く、表現性状も加味された細菌分類基準の導入が求められ、さらなる検討が行われる必要があります。
掲載論文
【題 名】 Inconsistent identification of Apilactobacillus kunkeei-related strains obtained by well-developed overall genome-related indices. Systematic and Applied Microbiology
【著者名】 Shintaro Maeno, Akihito Endo
【掲載誌】
Systematic and Applied Microbiology
【掲載DOI】10.1016/j.syapm.2024.126559.
謝辞
本研究は日本乳酸菌学会と公益財団法人発酵研究所(IFO) の助成を受けたものです。

