転写因子IRF8の発現量を調節する新たなエンハンサーが骨髄系細胞の分化運命を決定することを生体レベルで解明
横浜市立大学大学院医学研究科免疫学 村上 紘一 特任助手、佐々木 悠 博士研究員、西山 晃 准教授や田村 智彦 教授らの研究グループは、同 幹細胞免疫制御内科学、北里大学、米国国立衛生研究所と共同で、骨髄系細胞への分化の際に単球、樹状細胞、好中球のいずれになるかは転写因子IRF8の発現量の違いで決まることや、その分子メカニズムを明らかにしました。
研究成果のポイント
- 単核貪食細胞の分化に重要な転写因子IRF8をコードする遺伝子の発現を最も早期から誘導する新規のゲノム領域(エンハンサー)を発見した。
- このエンハンサーは上流の転写因子RUNX–CBFβによって駆動される。
- このエンハンサーを含む複数のエンハンサーによって調節されるIRF8の発現量の違いによって、骨髄系細胞(単球、樹状細胞、好中球)のどれが分化産生されるかの運命が決定される。
※本研究成果は、国際科学誌『Nature Immunology』に掲載されました(英国時間2月18日付:日本時間2月19日付オンライン)。
研究の背景
免疫を司る白血球は、大きく骨髄系とリンパ系に分けられます。前者には好中球をはじめとする顆粒球や、単球・マクロファージ・樹状細胞からなる単核貪食細胞(*1)が含まれ、後者にはB細胞、T細胞、NK細胞などが含まれます。これらの多様な細胞は、全て造血幹細胞(*2)が段階的に分化能を失った複数の前駆細胞段階を経て分化していくことで作り出されます。細胞分化の過程では、それぞれの細胞に固有の遺伝子発現パターンが確立されていきますが、この遺伝子発現の調節には、転写因子(*3)が結合するエンハンサー(*4)と呼ばれるゲノム領域が重要な役割を果たすことが知られています。
IRF8は単核貪食細胞の分化に必須の転写因子であり、IRF8が欠損すると単球や樹状細胞の産生が損なわれる一方で、著明な好中球の増加が引き起こされます。IRF8の発現は多能性造血前駆細胞(*5)の一部から始まり、単核貪食細胞の前駆細胞でさらに高まります。これが分化に重要な標的遺伝子のエンハンサーを準備・活性化し、最終的にそれらの下流の遺伝子群の発現を誘導して、単球・樹状細胞が産生されます。また、IRF8自体の発現も複数のエンハンサーによって調節されています。これまでに、生体内において分化の最終段階に近い細胞でIRF8発現を調節するエンハンサーは知られていましたが、早期の分化段階でIRF8の発現を担うエンハンサーは不明でした。さらに、単一の転写因子IRF8が単球と樹状細胞という異なる性質を持つ二つの細胞種の分化を支持するメカニズムも分かっていませんでした。
研究の内容
本研究では、まずIRF8の発現調節機構を明らかにするために、種々の分化段階の細胞をマウスの生体内から単離し、特にIrf8遺伝子座周囲に着目して、活性化エンハンサーの分布を次世代シーケンス技術(*6)で解析しました。その結果、これまでに報告されていたいずれのエンハンサーよりも遠位に新規のエンハンサー領域が存在することを発見しました。この領域は多能性造血前駆細胞の段階から活性化し始め、単核貪食細胞の前駆細胞へと分化が進むにつれて活性がさらに上昇していました。
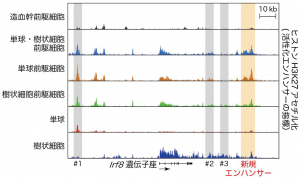
図1:Irf8遺伝子座周囲の活性化エンハンサーの分布
単核貪食細胞の各分化段階におけるIrf8遺伝子座周囲の活性化エンハンサーの分布を次世代シーケンス解析によって評価した。#1, #2, #3(灰色)は既に報告されていたエンハンサーだが、今回#3よりさらに遠位に新規エンハンサー領域(オレンジ)を見出した。
次に、この新たに見出したエンハンサー領域の生体内での働きを調べるために、ゲノム編集技術(*7)を用いてこのエンハンサー領域を欠損したマウスを作製しました。前述の通り、IRF8は多能性造血前駆細胞の段階から発現を開始し、単核貪食細胞系の細胞において高発現します。しかし、このエンハンサーを欠損すると、IRF8の発現量はほぼ全ての分化段階で著しく低下し、樹状細胞前駆細胞や樹状細胞(正確には抗ウイルス応答や抗腫瘍免疫に必須のcDC1と呼ばれる分画)が産生されなくなっていました。これはIRF8発現が完全に消失するIRF8欠損マウスと同様です。しかし意外にも単球については、IRF8欠損マウスとは反対に、著しく増加するという違いがありました。さらにIRF8の発現量に応じて協調あるいは拮抗する転写因子(それぞれPU.1・BATF3とC/EBP)が変化し、エピジェネティック(*8)な遺伝子発現制御作用が変化することで、好中球・単球・樹状細胞への分化運命を決定づけることが明らかになりました。なお本研究では既知のエンハンサー二つについても欠損マウスを作成し、解析しています。
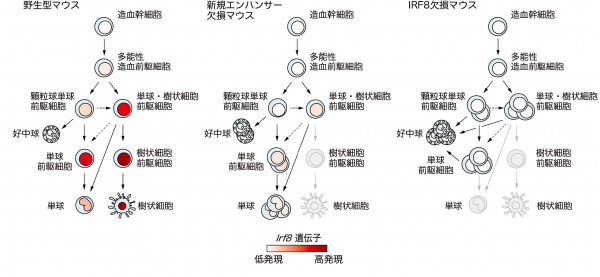
図2:新規Irf8エンハンサー欠損マウスにおける骨髄系細胞分化の変化
野生型マウス(左)では、造血幹細胞から多能性造血前駆細胞を経て、好中球・単球・樹状細胞が産生される。IRF8が全く発現しないIRF8欠損マウス(右)では、単球・樹状細胞への分化が障害され、その前駆細胞が蓄積する。それらの細胞も好中球へ分化するために著しい好中球の増加を認める。今回新たに発見したエンハンサーを欠損したマウス(中央)では、IRF8が発現を開始する多能性造血前駆細胞の段階から、産生される全ての骨髄系細胞においてIRF8の発現量が減少するがゼロにはならない。その結果、IRF8欠損マウスと同様に樹状細胞はその前駆細胞から産生が損なわれるが、IRF8欠損マウスとは逆に単球が増加する。
さらに、Irf8の新規エンハンサー領域を活性化する上流転写因子の検索を試みました。転写因子の結合部位は、クロマチン(*9)が開きDNAが露出した状態(オープンクロマチン)になっています。そこで、ATAC-seq(*10)というオープンクロマチン領域を全ゲノムで調べる技術を用いて、種々の分化段階を解析したところ、今回見出したIrf8の新規エンハンサー領域は造血幹細胞ではクロマチンが閉じた状態であり、多能性造血前駆細胞で初めてオープンクロマチンになることが分かりました。バイオインフォマティクス解析(*11)により、単核貪食細胞が分化する過程においてこのエンハンサーと同じパターンの活性化の推移を辿る領域に共通する特徴を見出しました。このエンハンサー領域は血球分化に重要な転写因子の一つであるRUNX–CBFβ複合体によって制御されることが明らかになりました。ヒトのIRF8遺伝子座の下流にもRUNX–CBFβによって制御される領域が存在しており、今回発見したIRF8の発現調節メカニズムとそれによる細胞分化の運命決定機構は生物種を超えて保存されている重要な仕組みである可能性が考えられます。
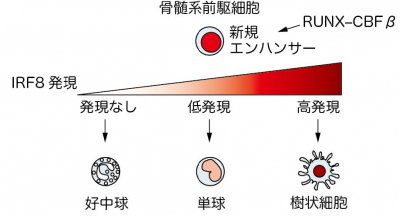
図3:前駆細胞でのIRF8発現量による骨髄系細胞の分化運命決定
骨髄系前駆細胞において、転写因子RUNX–CBFβによって駆動される新規エンハンサーがIRF8の発現を制御しており、その発現量の違いによって、細胞の分化はIRF8の発現がなければ好中球へ、低発現では単球へ、高発現では樹状細胞へと運命決定される。
今後の展開
今回の研究結果から、生体内での白血球分化では、複数あるIrf8エンハンサーはそれぞれ異なる分化段階や細胞系譜で作用することで、IRF8発現のタイミングや量を精密に制御していることが分かりました。そしてそのように調節されるIRF8の発現量が、どの種類の白血球を産生するかの決定に極めて重要であることが明らかになりました。このような細胞分化の運命決定メカニズムは、他の転写因子や細胞種でも見られる普遍的な仕組みであることが予想されます。
また、ヒトにおいてIRF8の発現量や活性の低下は、IRF8遺伝子自身の変異や慢性骨髄性白血病の原因遺伝子BCR-ABLなどによって引き起こされ、免疫不全や好中球増多症の原因となり得ます。加えてIrf8の上流転写因子であるRUNX–CBFβの遺伝子異常は、急性白血病などの血液がんの原因の一つとなることが知られています。したがって本研究の成果はこれらの病態解明・治療法の開発にも繋がると考えられます。
エンハンサーが遺伝子の発現制御に重要であることは広く認知されてきていますが、詳細な分子機構にはまだ不明な点が多いのが現状です。今後、Irf8遺伝子と複数のエンハンサー群がどのように相互作用して細胞分化に最適な遺伝子発現を導くかなどの解析を進め、細胞分化の基本原理を解明していきたいと考えています。
用語説明
*1 単核貪食細胞:
単球・マクロファージと樹状細胞の総称。これらの細胞は貪食能や抗原提示能などの機能を有しており、自然免疫と獲得免疫を繋ぐ重要な免疫細胞である。
*2 造血幹細胞:
骨髄内に存在し、全ての血液細胞に分化する能力と、細胞分裂しても自らを維持する自己複製能を有する組織幹細胞の一種。
*3 転写因子:
それぞれ特徴的なDNA配列を認識して様々なゲノム部位に結合することで、遺伝子からRNAの転写を制御するタンパク質。ヒトでは1,500種類以上あると言われている。
*4 エンハンサー:
遺伝子の転写開始点から離れた領域に存在し、転写因子が結合することで遺伝子の発現を調節するゲノムDNA領域。
*5 多能性造血前駆細胞:
骨髄に存在する細胞で、成熟した複数の血球細胞を作り出す能力を持つ。
*6 次世代シーケンス技術:
DNA断片の配列を並列的に極めて短時間で解析する技術。この方法を応用することで、全ての遺伝子の発現量を調べること(RNAシーケンス)や、エンハンサーの状態や転写因子の結合を全ゲノム規模で解析すること(クロマチン免疫沈降シーケンス)が可能となった。
*7 ゲノム編集技術:
DNA二本鎖を切断して、ゲノム配列の任意の領域を欠失させたり、他の配列と置換したり、外来遺伝子を挿入したりすることができる遺伝子編集技術。
*8 エピジェネティック:
DNA塩基配列の変化を伴わずにヒストンやDNAの化学修飾などによってクロマチンに作用すること。
*9 クロマチン:
ゲノムDNAとヒストンなどのタンパク質による複合体。クロマチンの主な構成分子は、芯となる8個のヒストンにDNAが約2周巻き付いたヌクレオソームである。
*10 ATAC-seq:
オープンクロマチン領域にDNAの修飾酵素が入りやすい性質を利用して、DNAに目印をつけることで、全ゲノム規模でオープンクロマチン領域を同定できる技術。クロマチン免疫沈降シーケンスなどと比較して、少数の細胞で解析可能な点が特徴の一つ。
*11 バイオインフォマティクス解析:
様々な生物学的データを情報科学によって解析する技術。
※本研究は、文部科学省、日本学術振興会、上原記念生命科学財団、日本血液学会による研究助成と、本学先端医科学研究センターが認定されている文部科学省共同利用・共同研究拠点「マルチオミックスによる遺伝子発現制御の先端的医学共同研究拠点」の支援を受けて行われました。
掲載論文
A RUNX–CBFβ-driven enhancer directs the Irf8 dose-dependent lineage choice between DCs and monocytes.
Koichi Murakami*, Haruka Sasaki*, Akira Nishiyama*, Daisuke Kurotaki, Wataru Kawase, Tatsuma Ban, Jun Nakabayashi, Satoko Kanzaki, Yoichi Sekita, Hideaki Nakajima, Keiko Ozato, Tohru Kimura, and Tomohiko Tamura (*Co-1st authors)
Nature Immunology, Feb 18, 2021, doi: 10.1038/s41590-021-00871-y
- 本件に関するお問合わせ先
-
横浜市立大学 広報室
E-mail:koho@yokohama-cu.ac.jp