がん治療薬の開発に向けたUHRF1の機能阻害剤を発見
横浜市立大学大学院生命医科学研究科 構造生物学研究室 有田 恭平 教授、郡 聡実さん(博士後期課程3年)、同研究科 生命情報科学研究室 浴本 亨 助教、柴橋 佑希 特任助手、池口 満徳 教授らを中心とした研究グループは、がん治療薬開発の標的となるタンパク質UHRF1 *1 の機能阻害剤を、構造生物学と計算科学を組み合わせた手法で発見しました。本成果はUHRF1を標的とした新たな作用点となるがんの薬剤開発の基盤となります。研究成果のポイント
構造生物学と計算科学を組み合わせ、UHRF1機能阻害剤となる化合物:5A-DMPを同定 5A-DMPとUHRF1との複合体構造を決定し、5A-DMPが標的部位に結合することを解明 5A-DMPがUHRF1とDNAリガーゼ1(LIG1*2 )の結合を阻害することを解明
研究背景 *3 異常があります。ヒトの体は数10兆個もの細胞で構成されていますが、細胞の種類としては筋肉細胞や神経細胞、血液細胞など約270に絞られています。たとえば、血液細胞のひとつである白血球としての構造と機能を個々の細胞に持たせるためには、それに相当する遺伝子の発現が必要になります。この遺伝子の発現を制御しているのがDNAメチル化であり、パターンによって、様々な細胞の種類が決まっています。DNAメチル化がヒトの生涯を通して維持されることで、細胞は正常に増殖することができます。これをDNA維持メチル化といいます。https://www.yokohama-cu.ac.jp/news/2017/20170811Arita.html )。https://www.yokohama-cu.ac.jp/news/2018/201901arita.html )。この立体構造の解明から、TTDドメインとLIG1の結合には、TTDドメインの「アルギニン結合溝」と名付けたポケットとLIG1のアルギニン121の相互作用が重要であることが分かりました。研究内容
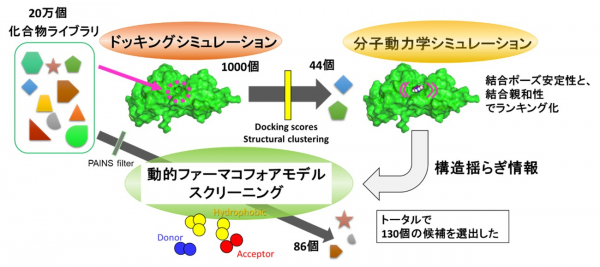
2019年に報告したUHRF1-TTDドメインとLIG1K126me3の複合体の結晶構造を基に、ドッキングシミュレーションを実施 TTDドメインのアルギニン結合溝に高い確率で結合すると予測された化合物に対しては、横浜市立大学が保有するスーパーコンピュータ上で分子動力学計算を行い、水分子やタンパク質の構造の揺らぎも指標に加えて評価 この分子動力学計算からTTDドメインのアルギニン結合溝と化合物の間で形成された相互作用情報を抽出し、その種類や頻度を反映させた動的ファーマコフォアモデル *4 を作成、当該モデルに当てはまる化合物をライブラリの中からさらに選出
図1 計算科学による候補化合物の選出の概略図
【構造生物学による化合物の結合様式の解明と機能阻害の検証】
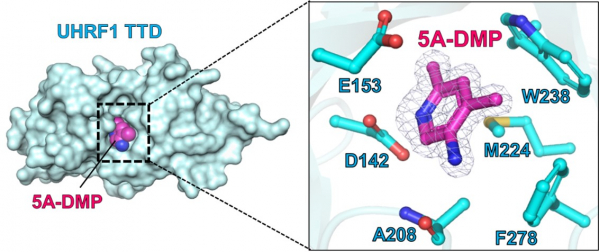
図2 TTDドメインと5A-DMPの複合体のX線結晶構造
さらに無細胞実験系を用い、5A-DMPがUHRF1とLIG1の結合を阻害できることも検証しました(東京大学 中西真 教授、西山敦哉 准教授との共同研究)。LIG1を特異的に認識する抗体を用いて、免疫沈降させた画分にUHRF1が含まれているかどうかで、UHRF1とLIG1の結合を評価しました。5A-DMPが存在しない時は、免疫沈降した画分にUHRF1が含まれており、UHRF1とLIG1が結合することが示されていますが、5A-DMPを加えるとその濃度依存的にUHRF1とLIG1の結合が見られなくなることが分かりました。このことから、今回発見した5A-DMPがUHRF1-TTDドメインのアルギニン結合溝に結合して、LIG1との結合を阻害する働きを持つことを明らかにしました。今後の展開 研究費 論文情報 用語説明 関連論文 http://dx.doi.org/10.1016/j.molcel.2017.07.012 https://doi.org/10.1016/j.str.2018.11.012